| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- ssh
- Jupyter notebook
- 오블완
- numpy
- cocre
- 히비스서커스
- docker attach
- HookNet
- Decision Boundary
- 기초확률론
- 도커
- vscode
- docker
- aiffel exploration
- docker exec
- cs231n
- WSSS
- IVI
- GIT
- AIFFEL
- 사회조사분석사2급
- Multi-Resolution Networks for Semantic Segmentation in Whole Slide Images
- logistic regression
- Pull Request
- 코크리
- 티스토리챌린지
- 프로그래머스
- 백신후원
- airflow
- CellPin
- Today
- Total
히비스서커스의 블로그
[Paper] HookNet: multi-resolution convolutional neural networks for semantic segmentation in histopathology whole-slide images 논문 정리 본문
[Paper] HookNet: multi-resolution convolutional neural networks for semantic segmentation in histopathology whole-slide images 논문 정리
HibisCircus 2021. 11. 3. 16:53※ Mart van Rijthovena, Maschenka Balkenhol , Karina SiliÅa, Jeroen van der Laak, Francesco Ciompi, 2021, HookNet: multi-resolution convolutional neural networks for semantic segmentation in histopathology whole-slide images의 논문을 읽고 제가 직접 이해한대로 정리해본 글입니다. 논문 내용이 길어 제가 중요하다고 생각하는 부분만 정리하였습니다. 잘못된 내용이 있다면 지적해주세요.
개인적인 정리
digital pathology에서 multi-resolution network로 제기되었던 MRN 모델에 이어 이보다 더 좋은 성능을 내었다고 하는 HookNet이 등장하였다. 기존 MRN에서는 context network의 encoder로 부터의 skip connection을 target network의 decoder 부분에 추가로 (크기를 키우는) 리사이즈 후 더해줌으로써 전체적인 feature를 더해주었다. 이에 발전하여 HookNet에서는 모든 부분에서 context network의 encoder로 부터의 skip connection이 아닌 한 곳에서의 skip connection을 target network의 decoder 부분에 맞게 잘래내어 더해줌으로써 (='hooking' mechanism) 전체적인 feature를 더해준다. 또한, context network에 대한 loss를 구하여 context network에 대한 학습도 이루지게 하였다. 이를 통해 MRN 보다 더 경량화되었음에도 더 좋은 성능을 내는 HookNet이 나오게 되었다.
Hooknet에서 사용한 데이터셋의 특징

breast tissue
- DCIS (ductal carcinoma in situ)
- IDC (invasive ductal carcinoma)
- ILC (invasive lobular carcinoma)
lung tissue
- TLS (tertiary lymphoid structures)
- GC (germinal centers)
1.2 The receptive field and field of view
receptive field
모델에 의해 관측가능한 입력의 확장영역. CNN의 receptive field는 filter size, pooling factor, convolutional and pooling layer 등에 의해 달라짐. 모델 최적화에서 절대적인 제한된 수가 적용됨. 모델 파라미터의 수의 감소 (feature map, mini-batch size, size of predicted output)
FoV(field of view)
관측정보를 고려하는 측면에서 field of view 개념이 존재한다. 입력 이미지 안에서 (픽셀이 드러내는 실제 공간)공간 위의 거리를 말한다. 그리고 입력이미지의 공간 해상도에 의존한다.
FoV는 receptive field에 대해 의미들을 갖는다.
면적 위의 거리가 함축되기 때문에 더 낮은 해상도의 이미지를 고려함으로써 같은 모델, 같은 사이즈, 같은 receptive field이더라도 더 넓은 FoV를 구성할 수 있다.
1.3 Multi-field-of-view multi-resolution patches
가능한 해상도에서 가장 높은 패치를 뽑아낼 때 아직은 전체적인 정보가 소실된 것은 아닌데 그 이유는 receptive field를 고려하지 않은 패치 주변에 이용가능한 조직들이 있기 때문이다.
같은 사이즈이나 다른 해상도를 가진 (WSI의 같은 위치의 중심에서 뽑은) 패치를 추출함으로써 같은 receptive field는 더욱 전체적인 정보(그리고 이전에 포함되지 않았던 정보)를 모을 수 있게 된다.
다른 해상도로 추출한 같은 사이즈의 중심이 같은 multiple patches는 multiple Field of View를 가진다고 해석할 수 있다. 이를 multi-field-view multi-resolution MFMR patches라고 할 수 있다.
클래스가 컨텍스트 및 세분화된 세부 사항에 따르는 것으로 알려진 다중 클래스 문제는 MFMR 패치 세트의 결합된 정보로부터 이익을 얻을 수 있다.
하지만, 이것은 여전히 해결되지 않은 문제이다.
문제는 고해상도로 탐지할 수 있는 미세한 세부 사항과 고정되지 않은 상황별 특징의 통합을 기반으로 고해상도 분할을 동시에 출력하는 것이다.
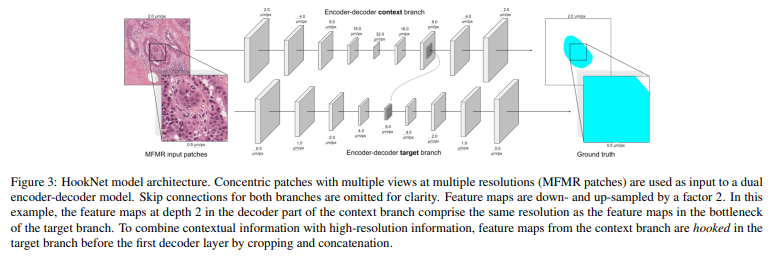
3. HookNet: multi-branch encoder-decoder network
3-1. Context and target branch
1) 두 개의 branch는 같은 구조를 가지나 가중치는 공유하지 않음
2) 두 개의 branch는 Unet을 기반으로 한 encoder와 decoder 모델임
3-2. MFMR input patches
HookNet의 input인 PC, PT는 (M x M x 3)의 크기를 가지며 다른 공간해상도를 가지는 한 쌍의 동심의 패치이다. 이러한 방식으로 Pt에 상응하는 FoV를 낮은 해상도의 Pc를 얻을 수 있다. 원활한 segmentation의 결과를 얻기 위해 그리고 인코더 디코더 branch안의 특징맵의 다르게 정렬되는 인공적인 원인을 피하기 위해 input patches의 사이즈와 해상도를 특정한 디자인 선택을 하였다.
1) encoder path안의 모든 특징맵은 각각의 pooling layer 전의 고른 크기를 가진 것과 같은 크기(= M)가 선택되어야 한다. Ronneberger et al. (2015)에서 언급되었듯 skip connections 뿐 아니라 branch를 가로지름에서도 특징 맵이 다르게 정렬되는 것이 발생할 수 있기 때문에 Hooknet에서는 이것이 매우 중요하다. 그러므로 이러한 제약은 특징 맵이 두 branch를 걸쳐 픽셀 단위로 정렬되어 남아있도록 해야한다.
2) rt(target patch의 해상도)와 rc(context patch의 해상도)는 디코딩 path 안의 특징맵의 쌍이 같은 해상도로 구성된 branch를 건너도록 하는 구조가 되도록 주어져야 한다. Hooking mechanism 참조
관행적으로 주어진 D의 값(인코더 디코더 구조의 깊이)으로부터 rT의 값과 rC의 값은 2DrT≥rC의 조건을 만족하도록 주어져야 한다.
3-3. Hooking mechanism
특징맵을 합치는 연산(concatenation)
- context branch와 traget branch path에서 추출한 특징맵을 concatenation
- Unet의 skip-connection의 효율을 보고 적용
- downstream layer가 모든 특징 맵에 걸쳐 작동하도록 허용
- 하이퍼 파라미터의 최적화 과정 중 작동 가능
hooking 부위
- target branch 안의 decoder의 시작부분으로 설정 (bottleneck layer를 거치기 전에 concatenate되면 안됨)
- 이는 concatenated된 특징맵이 target branch 안의 매 skip-connection으로부터 이점을 얻을 수 있는 곳
- 디코더 path안에서 내제된 up-sampling의 이점을 최대화할 수 있음
SRF
- spatial resolution of a feature map(특징맵의 공간적인 해상도)
- feature map에서 pixel-wise alignment를 보정하기 위해
- d: depth in the encoder-decoder model
- r: resolution of the input patch (μm/px=mpp)
- SRFCSRFT=2dC−dTrCrT=1 일 때, hooking mechanism을 적용가능
- 결과적으로 context branch에서의 central square region이 target brach에서의 feature맵과 상응하게 되는 결과
- 이는 이때 context branch와 target brach가 같은 해상도를 가지기 때문
- 이를 적용하기 위해 context branch에서 central square region 만큼 중심점에서 crop하여 target branch에 concatenate하는 것
3.4 Target and losses
HookNet의 목표는 PC와PT에 근거하여 segmentation map을 예측하는 것이다. 그러므로 target branch의 결과물로부터 계산되는 targetloss를 통해 backpropagated되는 single loss를 가져야 한다. 또한, context brach도 낮은 해상도의 패치를 예측하게 되고, 이 context error는 target loss와 동시에 구할 수 있다. 훈련의 목적으로 loss function을 L=λLhigh+(1−λ)Llow로 하였다. 여기서, Lhigh와 Llow는 target, context brach의 pixel-wise categorical cross entropy이며 λ는 각각의 branch의 중요도를 조절한다.
3.5 pixel-based-sampling
패치들은 sampling된 패치의 중앙 위치의 특정 조직 종류에 따라 sampling 된다. ground truth label의 날카로움 때문에 몇몇 패치들은 다른 패치들에 비해 더 적은 ground truth pixel을 가지게 된다. 훈련 과정 동안 모든 클래스 라벨이 pixel-based-sampling 전략에 근거하여 sampling 될 수 있도록 하였다.
첫번째 미니배치에서 패치들은 랜덤하게 뽑힌다. 다른 순차적인 미니배치에서 patch sampling은 이전의 미니배치에서 보여진 모든 클래스의 ground truth pixel의 축적에 근거하여 sampling한다. 축적된 픽셀의 양인 적은 클래스는 클래스에서 나타나지 않았던 것을 보정하기 위해 더 높은 가능성으로 추출한다.
3.6 Model training setup
patch
- 284x284x3 dimension
- mini-batch size 12
- L2 regularizer
model
- ReLU activation function
- 2x2 maxpooling and 2x2 nearest-neighbours
- soft activation function
loss
- λ : target branch에 대한 가중치 (= context branch에 대한 가중치는 1−λ)
training
- Adam optimizer
- learning rate 5∗10−6
- 200 epoches
4. experiments
resolution
0.5, 1.0, 2.0, 4.0, 8.0으로 하여 패치를 추출 후 학습하였음
model
Unet, MRN, HookNet
MRN과 HookNet의 차이점
| MRN | HookNet | |
| convolution | same convolution | valid convolution |
| encoder-decoder (archtecture) | a branch with an encoder only | using an additional brach consisting of an encoder-decoder |
| upsampling method | multiple independent upsampling | single upsampling via the decoder of the target branch |
| input size | 256x256x3 | 284x284x3 |
dataset
- breast dataset
- lung dataset
5. Results
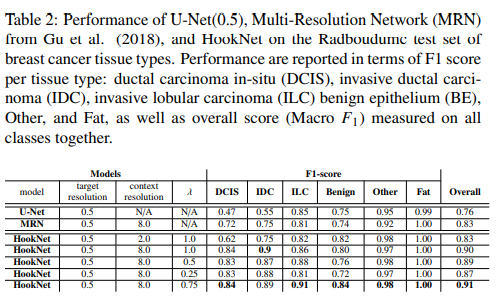
breast dataset
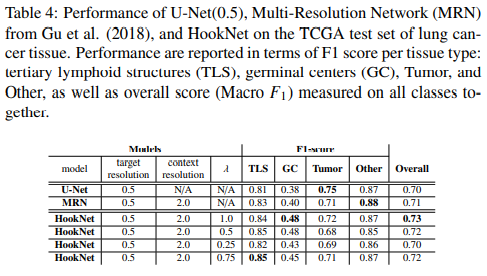
lung dataset
6. Disscusion
추가적으로 더 해보려는 시도
1. U-Net이 아닌 다른 encoder-decoder model를 적용해보려는 시도
2. histopathology image 뿐 아니라 nature image에 대해서도 적용해보려는 시도
3. field of view를 중간 부분을 포함할 수 있도록 (중배율을 포함할 수 있도록) 더 많은 branch를 통합해보는 시도
4. 더 좋은 GPU를 가지고 네트워크를 더 깊게 혹은 더 넓게 하든 inference의 시간을 줄이는 시도
5. 최적의 λ를 찾으려는 시도
6. sparse manual annotation이 되어있는 TCGA datsets과 Radboundumc의 데이터를 활용하였는데 좀 더 densely annotated data를 사용해보는 시도
-히비스서커스-




