| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- 코크리
- docker exec
- aiffel exploration
- vscode
- AIFFEL
- airflow
- numpy
- ssh
- Decision Boundary
- Multi-Resolution Networks for Semantic Segmentation in Whole Slide Images
- GIT
- 오블완
- docker attach
- HookNet
- Jupyter notebook
- WSSS
- 도커
- docker
- 히비스서커스
- cocre
- 티스토리챌린지
- Pull Request
- logistic regression
- 백신후원
- 사회조사분석사2급
- 프로그래머스
- 기초확률론
- CellPin
- IVI
- cs231n
- Today
- Total
히비스서커스의 블로그
[Cocre] 병리 이미지 다루기 1 (feat. Stain Normalization) 본문
이 글은 병리 이미지 다루기 시리즈 글입니다. 병리 이미지 다루기 1 (feat. Stain Normalization), 병리 이미지 다루기 2 (feat. Microns Per Pixel), 병리 이미지 다루기 3 (feat. Multi Resolution Model) 총 3회에 걸쳐 제작 예정입니다. 이 시리즈는 모두의 연구소의 코크리 2기 회원으로서 작성한 글임을 밝힙니다. 코크리란

안녕하세요. 저는 병원에서 병리 이미지 데이터로 딥러닝과 머신러닝을 적용하여 여러 과제들을 수행하는 연구하고 있습니다. 연구를 수행하면서 느끼는 점은 딥러닝 지식과 머신러닝 지식도 매우 중요하지만 도메인 지식도 꼭 필요하다는 것입니다. 당연한 이야기 이지만 결국 도메인 지식이 데이터를 이해하는데 도움을 주고 결과의 해석에 있어서도 중요하기 때문입니다. 'Garbage in, gargabe out'이라는 말도 이 부분에 적용할 수 있을 것 같네요. 그래서 이번에는 병리 이미지 특징을 살펴보도록 하겠습니다. 병리학에 관한 도메인 지식은 저도 부족하기에 데이터를 다룰 때 유의해야할 부분을 중심으로 정리하였으니 기겁하지 않으셨으면 좋겠습니다.
일반적으로 병리 이미지 데이터이라고 한다면 디지털화된 병리 슬라이드 이미지를 말합니다. 그럼 먼저 병리 슬라이드가 어떻게 생겼는지부터 살펴보겠습니다.

슬라이드 글라스 위에 얇게 썰은 병리조직을 올려놓고 커버 글라스로 덮어진 상태입니다. 오른쪽에 라벨 부분에는 연도나 환자의 정보를 간략하게 적어 보관합니다. 병리 슬라이드 제작 과정은 아래의 그림과 같습니다.

유의해서 보셔야 할 부분은 바로 염색 (staining)입니다. 병리 조직은 염색 과정 없이는 볼 수 없기 때문에 이 과정이 필요한데요. stain 방법으로는 H&E staining 방법과 immunohistochemistry staining 방법이 존재합니다. 일반적으로 H&E staining으로 염색을 하게 되면 붉은색과 보라색을 띠며 immunohistochemistry staining 방법은 푸른색과 갈색을 띠고 있습니다.

위의 그림과 같이 두 각각의 염색 과정 후 색상이 다르기에 병리 이미지 분석 시 분리하여 분석하는 것이 좋습니다. 일반적으로는 H&E staining 방법으로 염색을 많이 활용하는 것으로 알고 있습니다.
병리 이미지는 염색방법이 같더라도 언제하였는지 어느 병원에서 염색하였는지에 따라 배치 효과(batch effect)가 발생하여 차이가 존재합니다. 배치 효과란 일관된 작업을 처리하는 sample에 추가된 기술적인 변형의 차이가 나타나는 것을 말합니다. 이 차이는 병리 이미지 분석에서도 영향을 줄 수 있는데요. 이를 줄일 수 있는 방법은 잠시 뒤에서 살펴보겠습니다.
다음으로, 병리 슬라이드를 디지털화 하는 과정을 살펴보겠습니다. 병리 슬라이드는 아래 그림과 같이 디지털 스캐너를 통해서 스캔하여 디지털화합니다. 디지털 스캐너도 종류가 매우 다양합니다. Akoya, Olympus, Zeiss, KFBio, Leica, Philips 등등 이외에도 다양한 스캐너 회사가 존재합니다.
여기서 어떤 디지털 스캐너를 통해서 스캔을 했냐에 따라서도 색상의 차이가 존재합니다. 간단하게 동일한 병리 슬라이드에 대해 다른 스캐너로 스캔한 병리 영상 데이터에 대해 히스토그램을 살펴 RGB의 분포가 어느 값에 많이 위치하는지 살펴보겠습니다. 병리 이미지를 읽어들이기 위해서는 openslide 라이브러리를 사용해야 합니다.
import matplotlib.pyplot as plt
import numpy as np
import openslide
import glob
import cv2
color = ('b', 'g', 'r')
test_list = ['./Akoya.tiff', './KFBio.tiff', './Leica.tiff', './Philips.tiff', './Olympus.tiff', './Zeiss.tiff']
plt.figure(figsize=(10,30))
for j, test in enumerate(test_list):
slide = openslide.OpenSlide(test)
arr = np.array(slide.read_region((0,0), 0, size=slide.level_dimensions[0]))
w,h = slide.level_dimensions[0]
img = cv2.cvtColor(cv2.resize(arr, dsize=(w//10,h//10), interpolation=cv2.INTER_AREA), cv2.COLOR_BGR2RGB)
name = test.split('/')[-1][:-5]
plt.subplot(6,2,2*j+1)
plt.imshow(img)
plt.title(f'{name} Histogram')
for i, col in enumerate(color):
histr = cv2.calcHist([img], [i], None, [256], [0,256])
plt.subplot(6,2,2*j+2)
plt.plot(histr, color= col)
plt.xlim([0,256])
plt.title(f'{name} Histogram')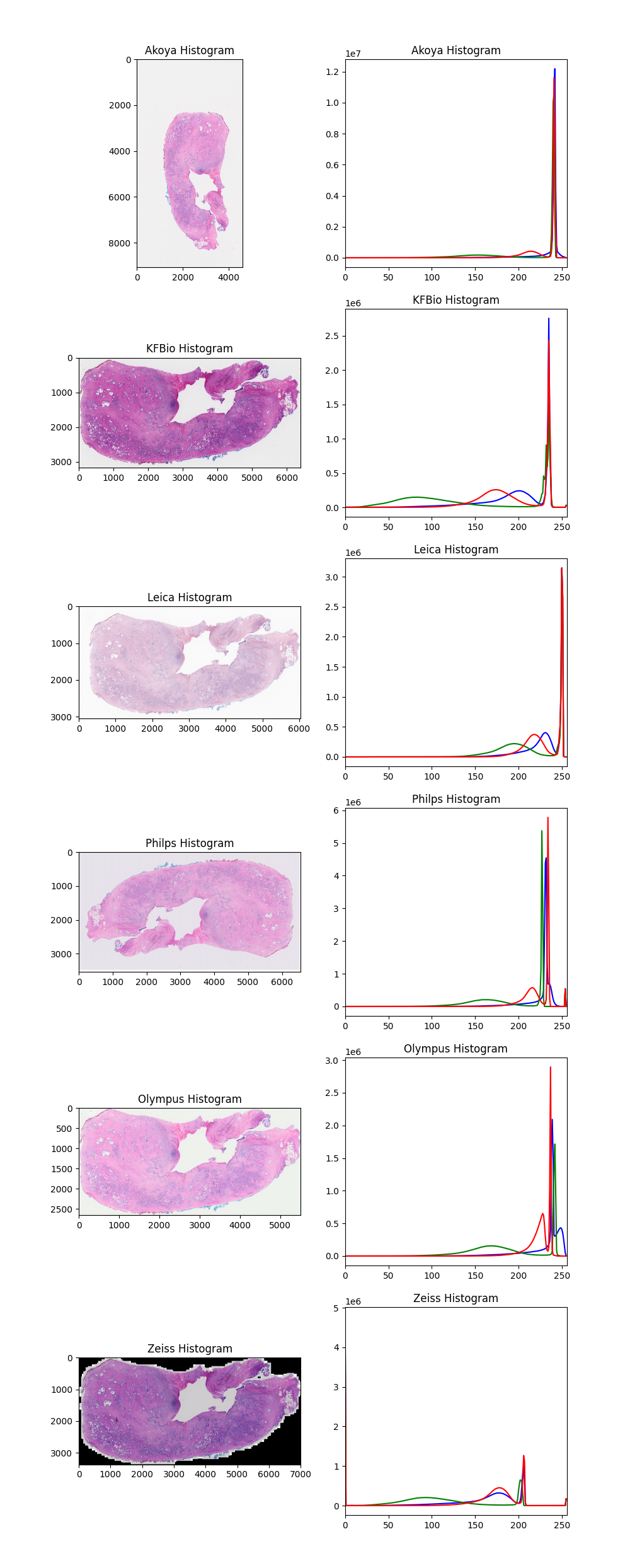
아까 염색 시에 배치 효과에 의하여 슬라이드 마다 차이가 발생할 수 있다고도 하였고 디지털 스캐너의 차이도 존재하니 이러한 차이들을 보정해주는 작업이 필요하겠죠? 이를 위해 보통 stain normalization의 작업을 해줍니다. staintools라는 패키지를 이용해보겠습니다.
혹시 staintools을 위해 SPAMS 설치 중 conda가 아닌 pip로 설치를 하시는 분들은 에러가 발생할 수 있는데요, 이때는 아래의 명령어를 입력해주시면 해결이 됩니다.
$ sudo apt-get install libatlas-base-dev liblapack-dev libblas-dev
병리 이미지를 분석 할 때에는 일정 크기의 패치 단위로 쪼개어서 분석합니다. stain normalization에서도 마찬가지입니다. 이에 관해서는 다음 시리즈에서 살펴보도록 하고 패치 이미지에 대해서 stain normailzation을 해보겠습니다. 먼저, 타겟 패치이미지와 적용 이미지를 불러옵니다. 적용 이미지 패치에서 색상의 분포를 가져와 타겟 이미지 패치의 색상 분포로 바꿔줍니다. 이때, 두 이미지 모두 표준화를 적용해준 다음 stain normailzation을 해주어야 합니다. 간단하게 Akoya를 적용 이미지 패치로 KFBio를 타겟 이미지 패치로 해보겠습니다.
import matplotlib.pyplot as plt
import staintools
import cv2
target = staintools.read_image('akoya_image_patch.png')
to_transform = staintools.read_image('kfbio_image_patch.png')
target_1 = staintools.LuminosityStandardizer.standardize(target)
to_transform_1 = staintools.LuminosityStandardizer.standardize(to_transform)
normalizer = staintools.StainNormalizer(method='vahadane')
target_2 = normalizer.fit(target)
transformed_2 = normalizer.transform(to_transform)
plt.figure(figsize=(15,10))
plt.subplot(2,3,1)
plt.imshow(target)
plt.subplot(2,3,2)
plt.imshow(target_1)
plt.subplot(2,3,4)
plt.imshow(to_transform)
plt.subplot(2,3,5)
plt.imshow(to_transform_1)
plt.subplot(2,3,6)
plt.imshow(transformed_2)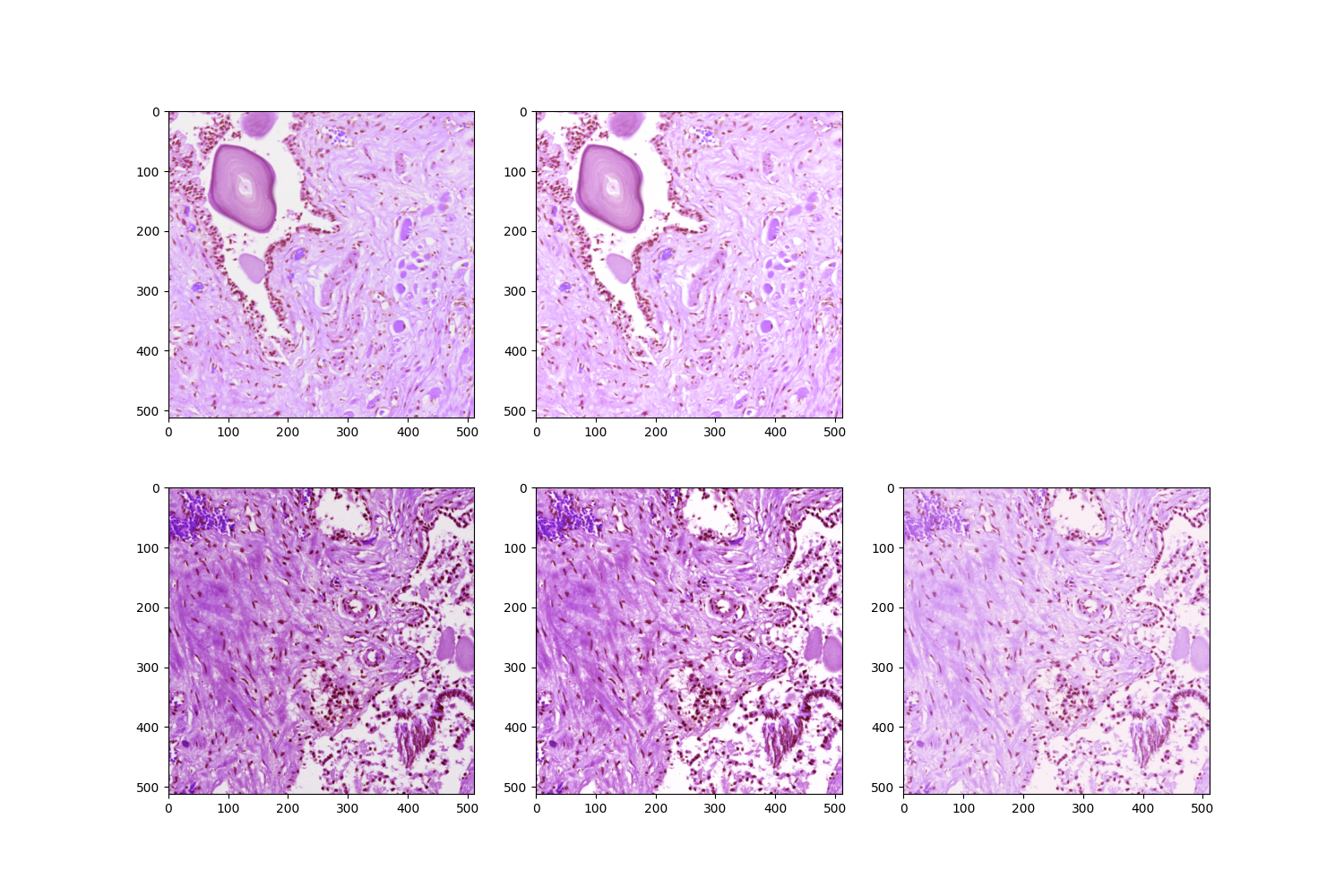
타겟 이미지 패치를 stain normalization을 해주니 적용 이미지 패치와 비슷한 색상을 가지게 되었으며 아래를 통해 분포도 유사해졌음을 확인할 수 있습니다.
import matplotlib.pyplot as plt
import cv2
import glob
test_list = [target_1, to_transform, transformed_2]
plt.figure(figsize=(10,15))
color = ('b', 'g', 'r')
for j, test in enumerate(test_list):
plt.subplot(3,2,2*j+1)
plt.imshow(test)
for i, col in enumerate(color):
histr = cv2.calcHist([test], [i], None, [256], [0,256])
plt.subplot(3,2,2*j+2)
plt.plot(histr, color= col)
plt.xlim([0,256])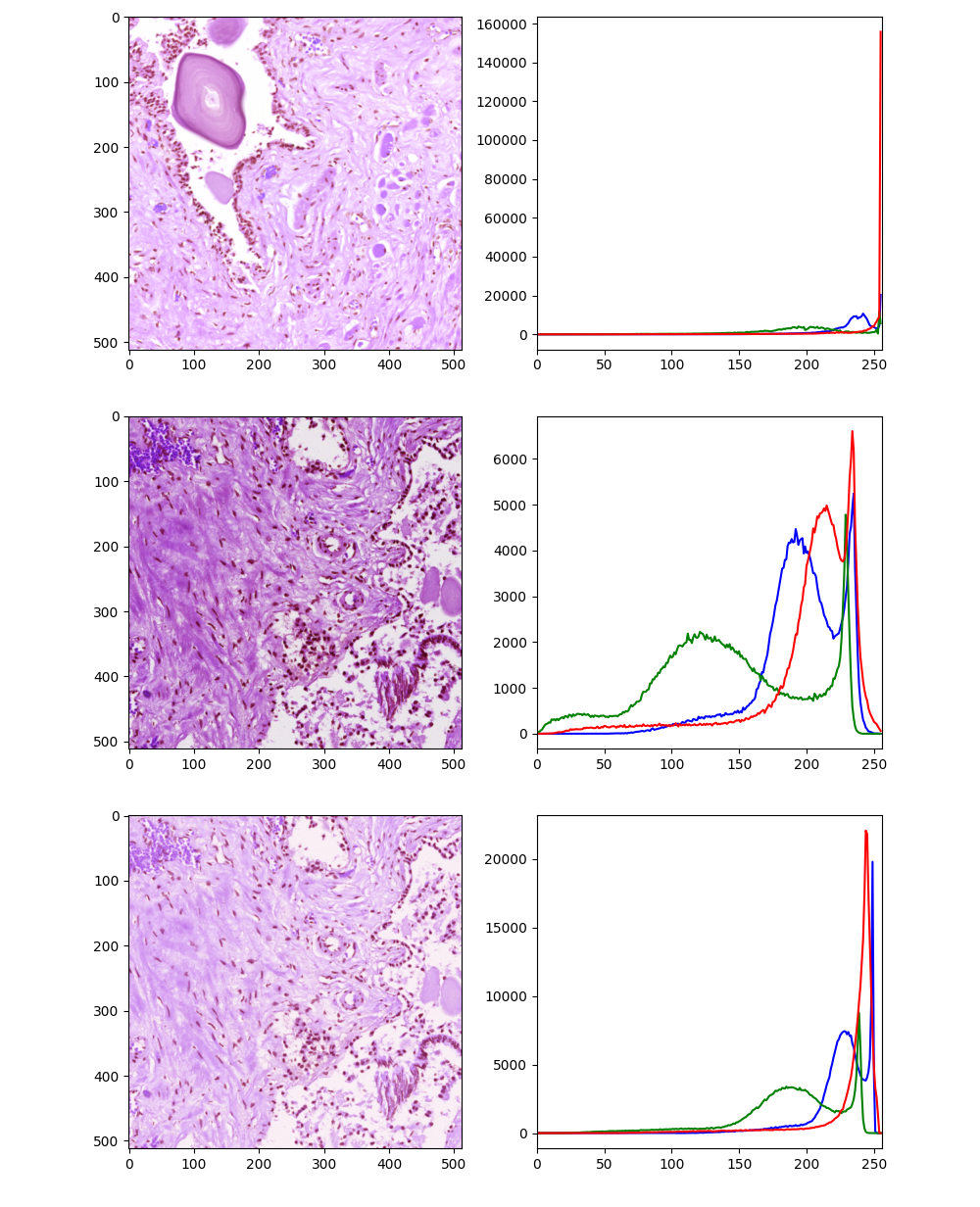
이와 같이 stain normalization을 통해 색상의 차이로 인한 오차를 줄여줄 수 있습니다. stain normalization을 통한 연구는 계속 진행되고 있으나 이는 병리 영상 데이터 분석 시 참고 사항일 뿐 필수적인 요소는 아닙니다. 다른 데이터들과 달리 병리 이미지는 색상이 중요한 특징 요소 중 하나인데 이에 대한 차이를 발생시키는 환경적 요인들과 극복할 수 있는 방법을 한 번 소개해드리고 싶었습니다.
병리 영상 데이터는 또 하나의 특징을 가지고 있는데요, 여러 개의 배율을 가진 이미지들이 하나의 파일에 담겨 있다는 것입니다. 위에서 slide.level_dimension 부분의 설명이 없었는데 이는 다음 편인 병리 이미지 다루기 2 - Slide Level (feat. Microns Per Pixels) 편에서 자세하게 알아보도록 하겠습니다. 읽어주셔서 감사합니다.




